分子生物学用ソフト virSEAKリポーティング用インポートNGS
お気に入りに追加する
商品比較に追加する
特徴
- 応用
- 分子生物学用
- 機能
- リポーティング用, インポート, NGS
- タイプ
- 自動
詳細
virSEAK (RUO - research use only) は、SARS-CoV-2の配列と利用可能な配列との比較を迅速かつ簡単に行うことができます。キットを使って個別に設定した後、fastqまたはfastaデータをロードするだけで、バリアントを含むアラインされた配列が得られます。
さらに、あなたの配列はセンザンコウ系統(例:B.1.1.7)およびGISAIDクレード(例:L)に割り当てられます。
また、virSEAKは、あなたの配列がスパイクタンパク質の変異体N501YとE484Kを含んでいるかどうかを自動的にチェックします。
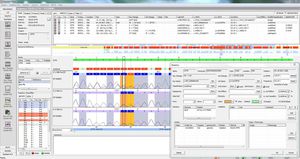
CoverageとQualityの値が表示されます:required-, absolute-, average- and median coverage, % of "wild type" and "N" bases.品質インジケータは、品質が定義された品質しきい値を満たしているかどうかを示します。
また、自動インポートおよびエクスポート機能とカスタマイズ可能な配列の転送により、ハイスループットNGSシーケンスに最適化されたツールとなっています。マルチファスタファイルを簡単に作成し、疫学的状況や変異の拡散を監視するための中央報告機関(例:ドイツはRobert Koch Institut)への報告を改善することができます。
virSEAK - インストール可能なバージョンです:
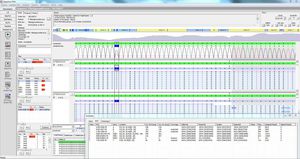
生のfastqまたは前処理済みのfastaシーケンスファイルを簡単にインポートできます。
自動化されたバッチインポートとエクスポートによるハイスループットNGSシーケンサー
N501YおよびE484Kの変異が含まれる場合、自動的に警告を発します。
Pangolinの系統とGISAIDのクレードに割り当てられる
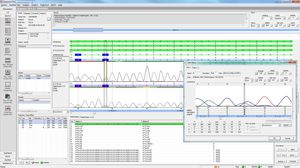
度数で他の変種を確認する
サブタイプ、所見数、所見位置の比較
配列とバリアントをエクスポート(メタ情報付き、fastaやcsvフォーマットで)。
結果が保存される
カスタマイズ可能なシーケンス転送による全自動ハイスループット・シーケンス
---
カタログ
この商品のカタログはありません。
JSI medical systemsの全カタログを見る