- 研究室
- 分子生物学
- 実験用ソフトウェアモジュール
- JSI medical systems
NGSシーケンシングソフトウェアモジュール SEQNEXT実験用分析キャプチャー
お気に入りに追加する
商品比較に追加する
特徴
- 応用
- 実験用, NGSシーケンシング
- 機能
- 分析, キャプチャー, 製図用
詳細
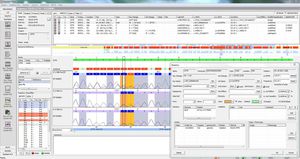
SEQNEXTは、次世代シーケンサーデータのマッピング、アライメント、バリアント検出のためのパワフルでユーザーフレンドリーなアプリケーションです。SEQNEXTは、一般的なシーケンスプラットフォームやキット(PCRベース、エンリッチメント、シーケンスキャプチャー)のデータを解析することができます。検出されたすべてのバリアント(長さを問わない欠失、挿入、インデル、SNP、リピート領域、CNV、遺伝子融合など)をわかりやすく直感的に視覚化することができます。
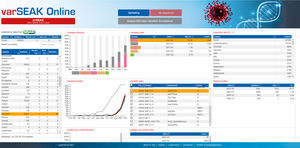
SEQNEXTは、dbSNP、1000 Genomes、ClinVar、ExAC、gnomADといった公開SNPデータベースにアクセスし、分類やフィルタリングを行います。すべての結果データは、経験や知識を共有するためのバリアントデータベース(varSEAK)と交換することができ、ラボの内部LIMシステムに転送したり、パーソナライズされた患者レポートとして発行したりすることができます。
NEW SARS-CoV-2などのウイルスシークエンスデータの解析を改善しました。
トリオアナリシスと改良型プールアナリシス
バリアントテーブルのSequence Ontology Annotationsについて
varSEAKとdbSNP, ClinVar, UK10K, ESP, gnomAD, ExACのような利用可能なすべての外部DBへのジャンプオプション。
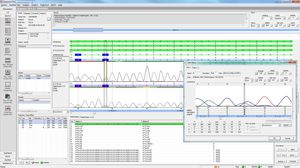
ホールエクソームシーケンス(WES)解析
すべての一般的な次世代シーケンスプラットフォームおよびキットのデータに対応しています。
hg19および/またはhg38に基づくベッドまたはマニフェストファイルのインポートにより、個々のターゲット領域(ROI)を簡単にセットアップ可能
WESデータのフィルタリングと解析のための遺伝子リストのインポート
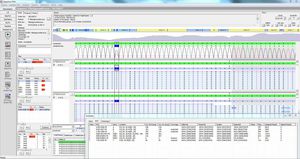
fastqファイル、fastq.gzファイル、bamファイルの解析
マッピング、アライメント、品質、バリアントコールの効率的な標準(使用アルゴリズム:BWA、Smith-Waterman、JSIで適応済み)
解析とバリアントコールのパーソナライズ設定(≥0.1 %)。
あらゆる長さの欠失、挿入、インデル、SNP、リピート領域、CNV、遺伝子融合などを高感度かつ特異的に検出可能
---
カタログ
SEQUENCE PILOT
2 ページ