NGSシーケンシングソフト NextGENe®分析実験用
お気に入りに追加する
商品比較に追加する
特徴
- 機能
- 分析
- 応用
- NGSシーケンシング, 実験用
詳細
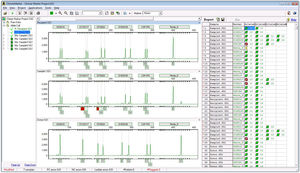
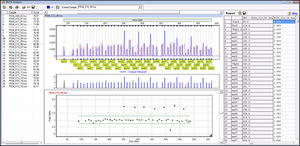
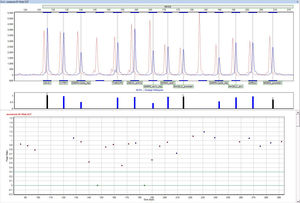
NextGENeソフトウェアは、Illumina® iSeq, Miniseq, MiSeq, NextSeq, HiSeq, NovaSeqシステム、Ion Torrent Ion GeneStudio S5, PGM, Protonシステム、その他のプラットフォームで生産されたデスクトップ型シーケンスデータの解析に最適な分析パートナーです。
NextGENeはWindows®オペレーティングシステム上で動作し、生物学者にとって使いやすい「ポイント&クリック」インターフェースを提供します。CLCゲノミクスワークベンチ、LasergeneのSeqMan Pro、MAQ & SOAP、Top Hat、BWA & Bowtieなどのアカデミックソフトウェアを使用する際に必要となるスクリプトや他のバイオインフォマティクスサポートは必要ありません。
NextGENeソフトウェアは、独自のプラットフォーム別技術を採用し、1つの独立したマルチアプリケーションパッケージです。NextGENeソフトウェアには、以下のような解析モジュールが含まれています:
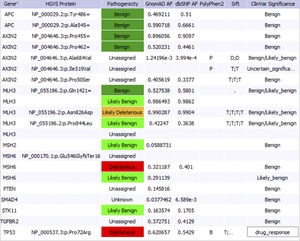
SNP/INDEL検出
ジャームライン
ソマティック
エクソームキャプチャ、ホールエクソームシーケンス(WES)
ホールゲノムシーケンス(WGS)
構造変異解析(融合遺伝子検出を含む)
ファミリーベースとトリオの比較
腫瘍と正常の比較
コピー数変異(CNV)検出とMLPA的スクリーニングの代替手段
デノボアセンブリ
トランスクリプトーム、代替スプライシング解析、トランスクリプト発現レベル
ChIP-Seq、デジタル遺伝子発現(DGE)、miRNA解析・定量化、メタゲノミクス
NextGENeは、生物学者に優しいWindows®環境で設計されており、バイオインフォマティクスのリソースやコストを大幅に削減することができます。NextGENeは、低コストな64ビットWindows OSベースのハードウェアを使用しています。(推奨ハードウェア構成はこちら)
---