- Laboratório
- Biologia molecular
- Software de criação de laudos
- JSI medical systems
Software de criação de laudos virSEAKde importaçãoNGSpara biologia molecular
Guardar nos favoritos
Comparar
Características
- Função
- de criação de laudos, de importação, NGS
- Aplicações
- para biologia molecular
- Tipo
- automático
Descrição
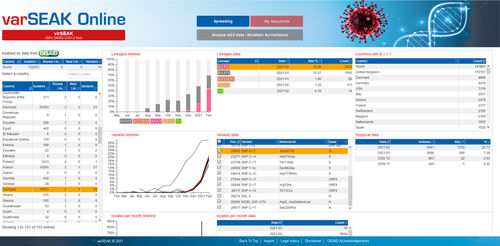
virSEAK (RUO - utilização apenas para investigação) oferece uma comparação rápida e conveniente da sua sequência SARS-CoV-2 com as sequências disponíveis. Após uma configuração personalizada com o seu kit, basta carregar dados fastq- ou fasta e obter uma sequência alinhada com as variantes.
Além disso, a sua sequência será atribuída a uma linhagem de pangolins (por exemplo B.1.1.7) e a uma clade GISAID (por exemplo L).
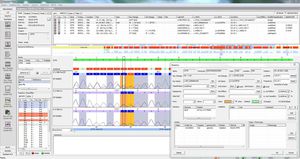
virSEAK também verifica automaticamente se a sua sequência contém as variantes da proteína spike N501Y e ou E484K.
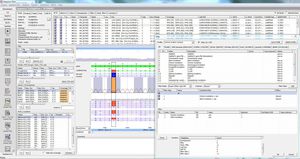
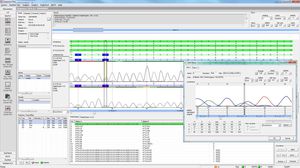
São mostrados valores de Cobertura e Qualidade, incluindo: cobertura requerida, absoluta, média e mediana, % de bases "tipo selvagem" e "N". Um indicador de qualidade mostra se a qualidade satisfaz os limiares de qualidade definidos.
Além disso, a ferramenta é optimizada para a sequenciação NGS de alto rendimento com funções automatizadas de importação e exportação e transferência de sequência personalizável. Criar facilmente ficheiros multi-fasta para melhorar a apresentação de relatórios a um gabinete central de relatórios para vigilância da situação epidemiológica e propagação de mutações (por exemplo, para a Alemanha, o Robert Koch Institut).
virSEAK - versão instalável:
importação fácil de ficheiros em bruto de sequência rápidaq ou fasta pré-processada
sequenciação NGS de alto rendimento com importação e exportação automática de lotes
alerta automático se a(s) mutação(ões) N501Y e/ou E484K estiver(em) contida(s)
obter uma atribuição a uma linhagem Pangolin e a um clade GISAID
verificar outras variantes com frequências
comparar subtipos, contagens e localizações dos resultados, ...
sequência e variantes de exportação (com meta-informação, em formato fasta e/ou csv)
os resultados são armazenados
sequenciação de alto rendimento totalmente automatizada com transferência de sequência personalizável
---
Catálogos
Não estão disponíveis catálogos para este produto.
Ver todos os catálogos da JSI medical systemsPesquisas relacionadas
- Software de análise
- Software de criação de laudos
- Software automático
- Módulo de software
- Software de importação
- Software servidor
- Módulo de software de análise
- Software para biologia molecular
- Módulo de software de laboratório
- Módulo de software de captura
- Módulo de software de controle
- Software NGS
- Módulo de software de mapeamento
* Os preços não incluem impostos, transporte, taxas alfandegárias, nem custos adicionais associados às opções de instalação e de ativação do serviço. Os preços são meramente indicativos e podem variar em função dos países, do custo das matérias-primas e das taxas de câmbio.