- Produtos
- Módulo de software de análise
- JSI medical systems
Módulo de software para sequenciamento SEQPATIENTde análisede laboratório
Guardar nos favoritos
Comparar
Características
- Função
- de análise
- Aplicações
- para sequenciamento, de laboratório
Descrição
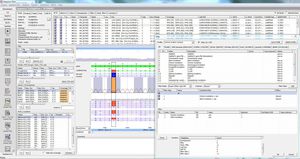
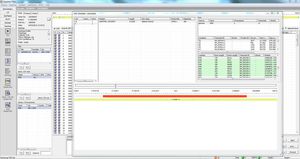
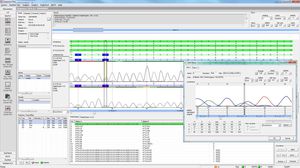
O SEQPATIENT é uma aplicação poderosa e de fácil utilização para alinhamento e detecção de variantes de dados de sequenciamento de Sanger. O SEQPATIENT pode analisar dados de todas as plataformas de sequenciação comuns. A visualização de todas as variantes detectadas, como eliminações, inserções, indels e SNPs, é clara e intuitiva. A análise de DNA genómico e cDNA é possível. Uma função estatística de área de pico garante a detecção de mutações de baixa frequência, tais como desistências alélicas, mosaicos e variantes somáticas.
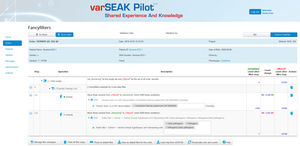
A SEQPATIENT oferece acesso a bases de dados públicas SNP como dbSNP, 1000 Genomes, COSMIC, ClinVar, ClinVitae, ExAC e gnomAD para classificação e filtragem. Todos os dados de resultados podem ser trocados com a nossa base de dados de variantes para Experiência e Conhecimento Partilhados - varSEAK, transferidos para sistemas LIM internos de laboratório e/ou emitidos como relatórios personalizados de pacientes.
Compatível com dados de todas as plataformas de sequenciação Sanger comuns
Fácil configuração de regiões-alvo individuais através da importação de ficheiros txt ou bed-files baseados em genes únicos descarregados (ENSEMBL / NCBI) ou hg19 / hg38
Chamador de base configurável com limiares dependentes do sequenciador
Corte automático e definido pelo utilizador de ficheiros de resultados de sequenciação
Identificação e genotipagem de doentes através de identificações SNP
Todos os resultados para um paciente apresentados num ecrã incl. variantes encontradas, alterações de aminoácidos, nomenclaturas HGVS, etc.
Alta sensibilidade e especificidade para a detecção de SNPs, supressões, inserções e indels
Base de dados interna de mutações mantendo o histórico de detecção de mutações do SEQPATIENT e SEQNEXT
---
Catálogos
SEQUENCE PILOT
2 Páginas
* Os preços não incluem impostos, transporte, taxas alfandegárias, nem custos adicionais associados às opções de instalação e de ativação do serviço. Os preços são meramente indicativos e podem variar em função dos países, do custo das matérias-primas e das taxas de câmbio.